複数の変数の分布を一度に確認したい場合があります。このような場合、tidy data形式を上手く使うと比較的簡単に比較用のプロットを描くことができます。
Packages and Datasets
本ページではR version 3.6.1 (2019-07-05)の標準パッケージ以外に以下の追加パッケージを用いています。
| Package | Version | Description |
|---|---|---|
| ggplot2 | 3.2.1 | Create Elegant Data Visualisations Using the Grammar of Graphics |
| GGally | 1.4.0 | Extension to ‘ggplot2’ |
| tidyverse | 1.2.1 | Easily Install and Load the ‘Tidyverse’ |
また、本ページでは以下のデータセットを用いています。
| Dataset | Package | Version | Description |
|---|---|---|---|
| iris | datasets | 3.6.1 | Edgar Anderson’s Iris Data |
| mtcars | datasets | 3.6.1 | Motor Trend Car Road Tests |
| wbcd | N/A | N/A | Breast Cancer Wisconsin (Diagnostic) Data Set, UCI |
複数のグラフで確認する
散布図行列は二変数間のデータ分布を比較するのに便利なグラフです。GGallyパッケージは二変数の関係のみならず各変数の分布も変数の型(連続変数、離散変数)に応じて可視化できます。例えばirisデータセットであれば下図のように可視化されます。
iris %>%
GGally::ggpairs(progress = FALSE, ggplot2::aes(colour = Species, alpha = 0.5))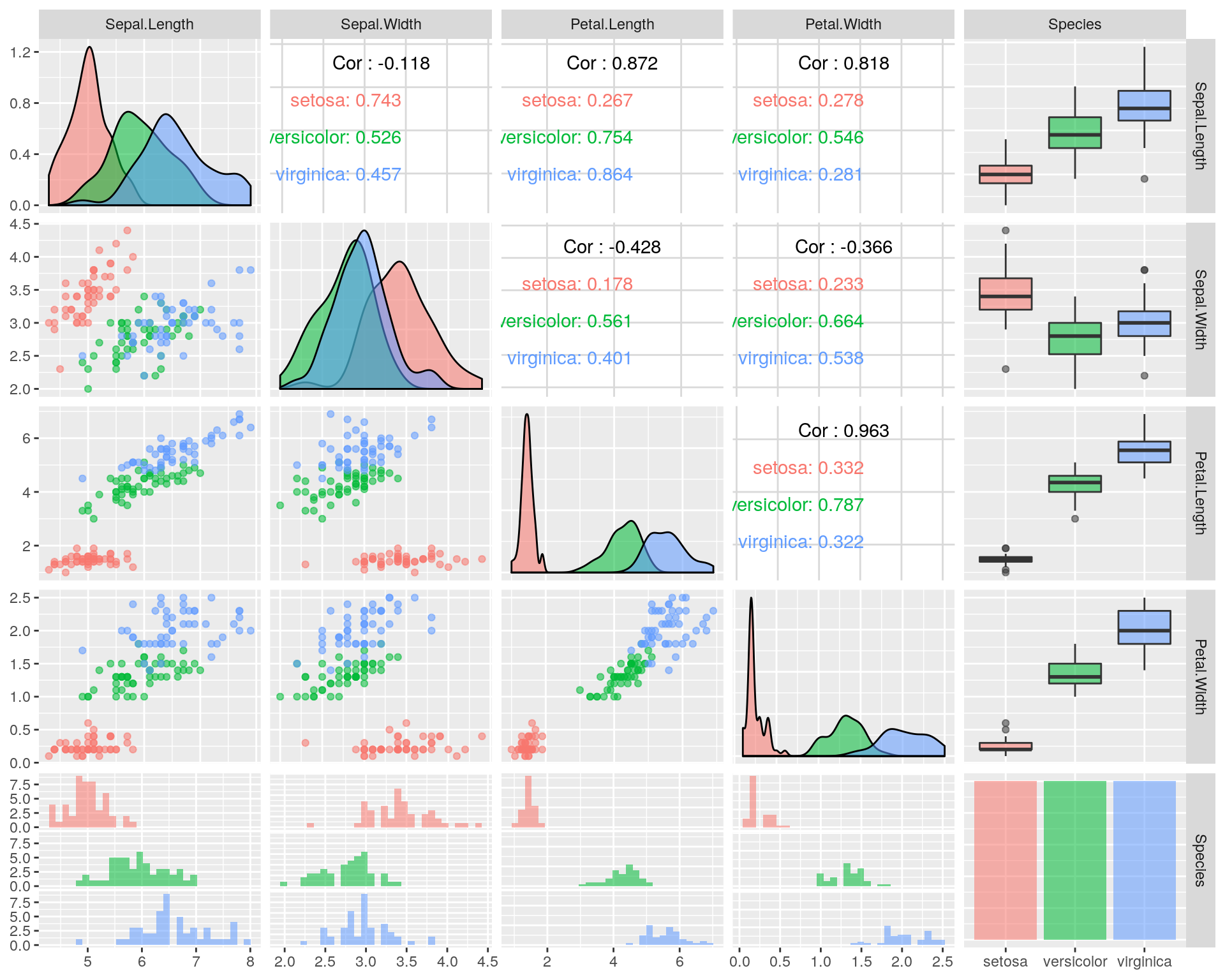
具体的な使い方は モダンな散布図行列 をご覧ください。
単一のグラフで比較する
単一のグラフ(例えば箱ひげ図)で各変数のデータ分布を比較したい場合には、元のデータをほんの少し変形することで比較が可能になります。
データの変形
irisデータセットは下表のような形式のデータセットです。
iris| Sepal.Length | Sepal.Width | Petal.Length | Petal.Width | Species | |
|---|---|---|---|---|---|
| 1 | 5.1 | 3.5 | 1.4 | 0.2 | setosa |
| 2 | 4.9 | 3 | 1.4 | 0.2 | setosa |
| 3 | 4.7 | 3.2 | 1.3 | 0.2 | setosa |
| … | … | … | … | … | NA |
| 148 | 6.5 | 3 | 5.2 | 2 | virginica |
| 149 | 6.2 | 3.4 | 5.4 | 2.3 | virginica |
| 150 | 5.9 | 3 | 5.1 | 1.8 | virginica |
このデータセットを更にtidyにまとめます。具体的には4列ある数値変数の変数名をvarsに、その値をvalueにまとめます。このようにすることで元からある因子型変数のSpeciesに加えて元の変数名を水準にもつvarsという因子型変数が作れます。
iris %>%
tidyr::gather(key = "vars", value = "value", -Species) | Species | vars | value | |
|---|---|---|---|
| 1 | setosa | Sepal.Length | 5.1 |
| 2 | setosa | Sepal.Length | 4.9 |
| 3 | setosa | Sepal.Length | 4.7 |
| … | NA | NA | … |
| 598 | virginica | Petal.Width | 2 |
| 599 | virginica | Petal.Width | 2.3 |
| 600 | virginica | Petal.Width | 1.8 |
この形式にすることでグラフ内をSpecies変数による層別(水準別)表示し、グラフ間をvars変数による層別(水準別)表示することができます。
iris %>%
tidyr::gather(key = "vars", value = "value", -Species) %>%
ggplot2::ggplot(ggplot2::aes(x = Species, y = value, colour = Species)) +
ggplot2::geom_boxplot() +
ggplot2::facet_wrap(~ vars)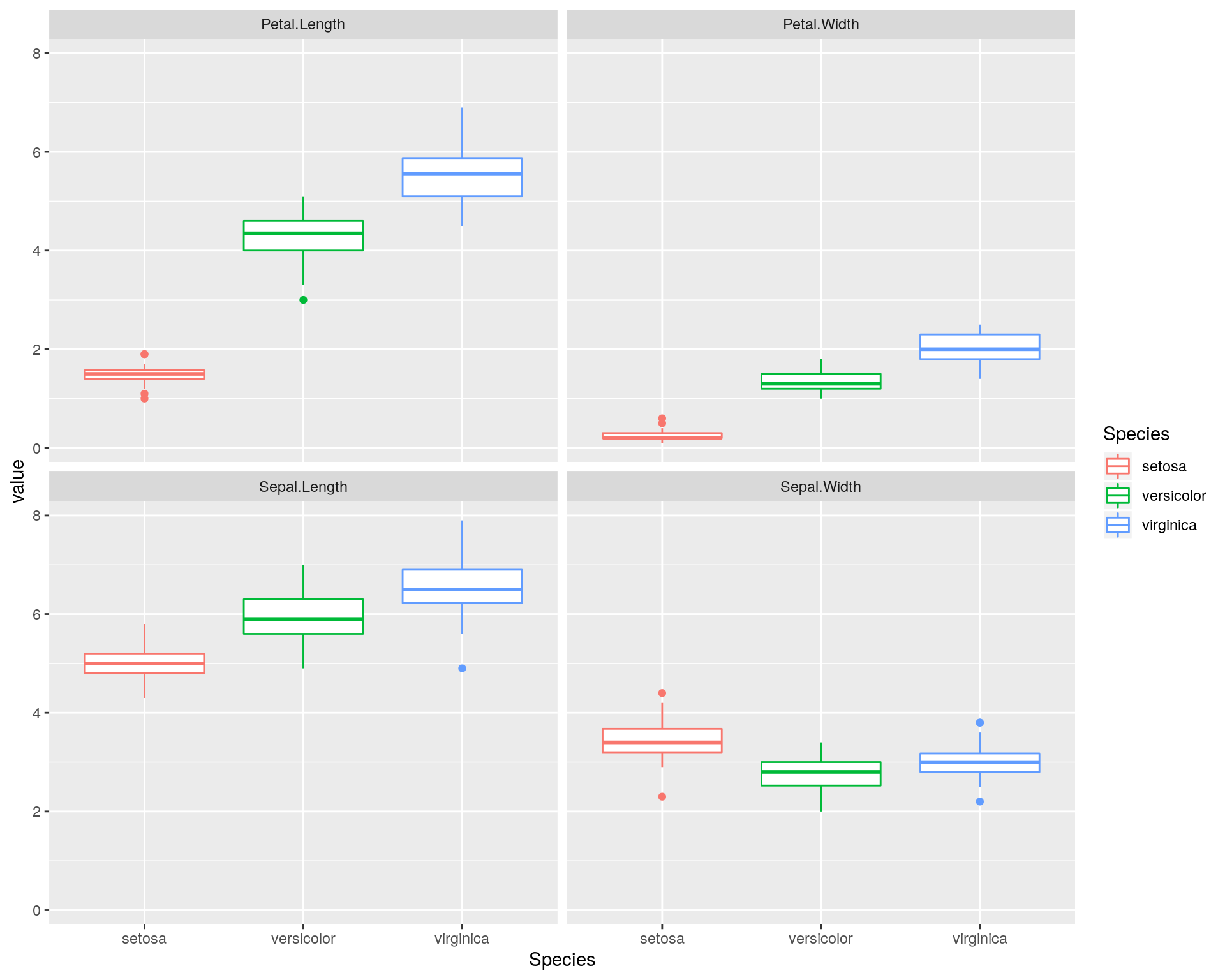
変数毎のレンジが大きく異なる場合
irisデータセットは、各変数の取る範囲が似たような範囲ですので上記のように同一スケールで描画しても特に問題はありません、しかしながら、mtcarsデータセットのように各変数の取る範囲が大きく異なる(2桁以上異なる)場合には下図のようになってしまい、比較することが困難になります。
mtcars %>%
dplyr::mutate(am = factor(am, labels = c("AT", "MT"))) %>%
dplyr::select(am, mpg, disp, hp, drat, wt, qsec) %>%
tidyr::gather(key = "vars", value = "value", -am) %>%
ggplot2::ggplot(ggplot2::aes(x = am, y = value, colour = am)) +
ggplot2::geom_boxplot() +
ggplot2::facet_wrap(~ vars)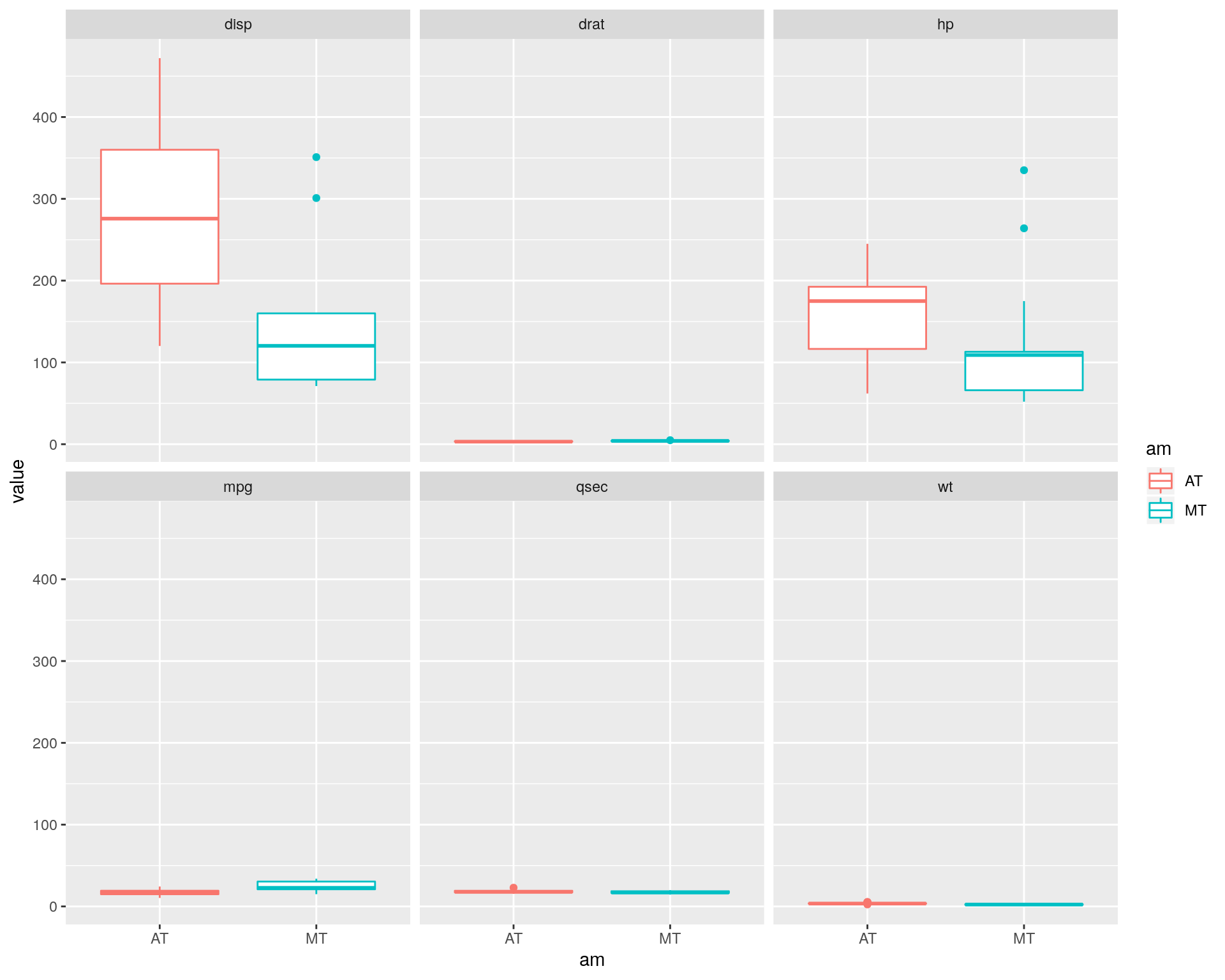
このような場合にはggplot2::facet_wrap関数にscalesオプションを指定してください。
mtcars %>%
dplyr::mutate(am = factor(am, labels = c("AT", "MT"))) %>%
dplyr::select(am, mpg, disp, hp, drat, wt, qsec) %>%
tidyr::gather(key = "vars", value = "value", -am) %>%
ggplot2::ggplot(ggplot2::aes(x = am, y = value, colour = am)) +
ggplot2::geom_boxplot() +
ggplot2::facet_wrap(~ vars, scales = "free_y")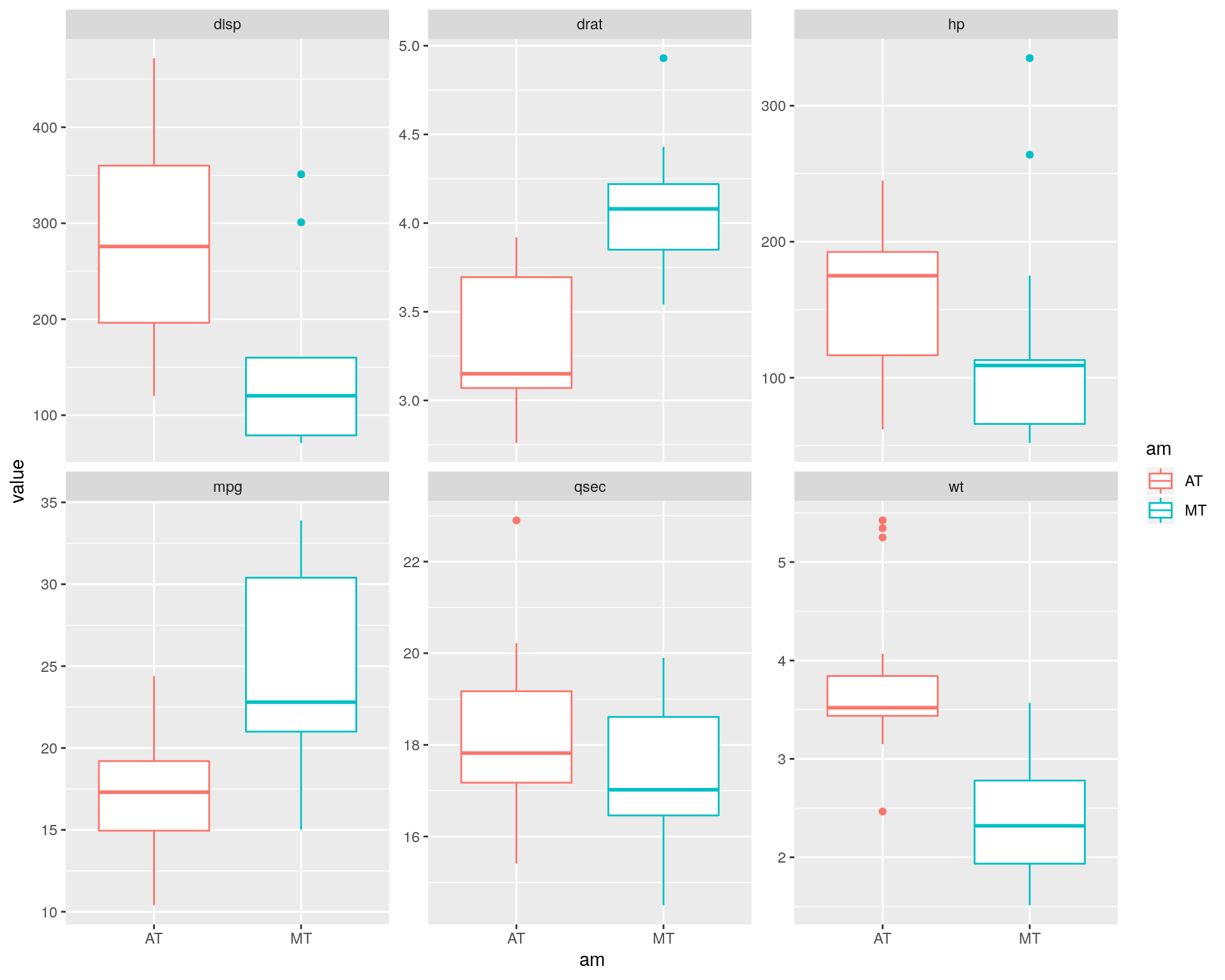
ヒストグラムで比較してみる
ggplot2::geom_boxplot関数の指定を変更すれば別のグラフで分布を比較することが可能です。例えばヒストグラムで比較したい場合にはggplot2::geom_histogram関数を指定します。ggplot2::box_plot関数と異なりX軸、Y軸共に取りうる値が変数毎にことなる可能性がありますのでggplot2::facet_wrap関数のscalesオプションを適宜変更してください。
iris %>%
tidyr::gather(key= "vars", value = "value", -Species) %>%
ggplot2::ggplot(ggplot2::aes(x = value,
fill = Species, colour = Species)) +
ggplot2::geom_histogram(position = "identity", bins = 10, alpha = 0.25) +
ggplot2::facet_wrap(~ vars, scales = "free_x")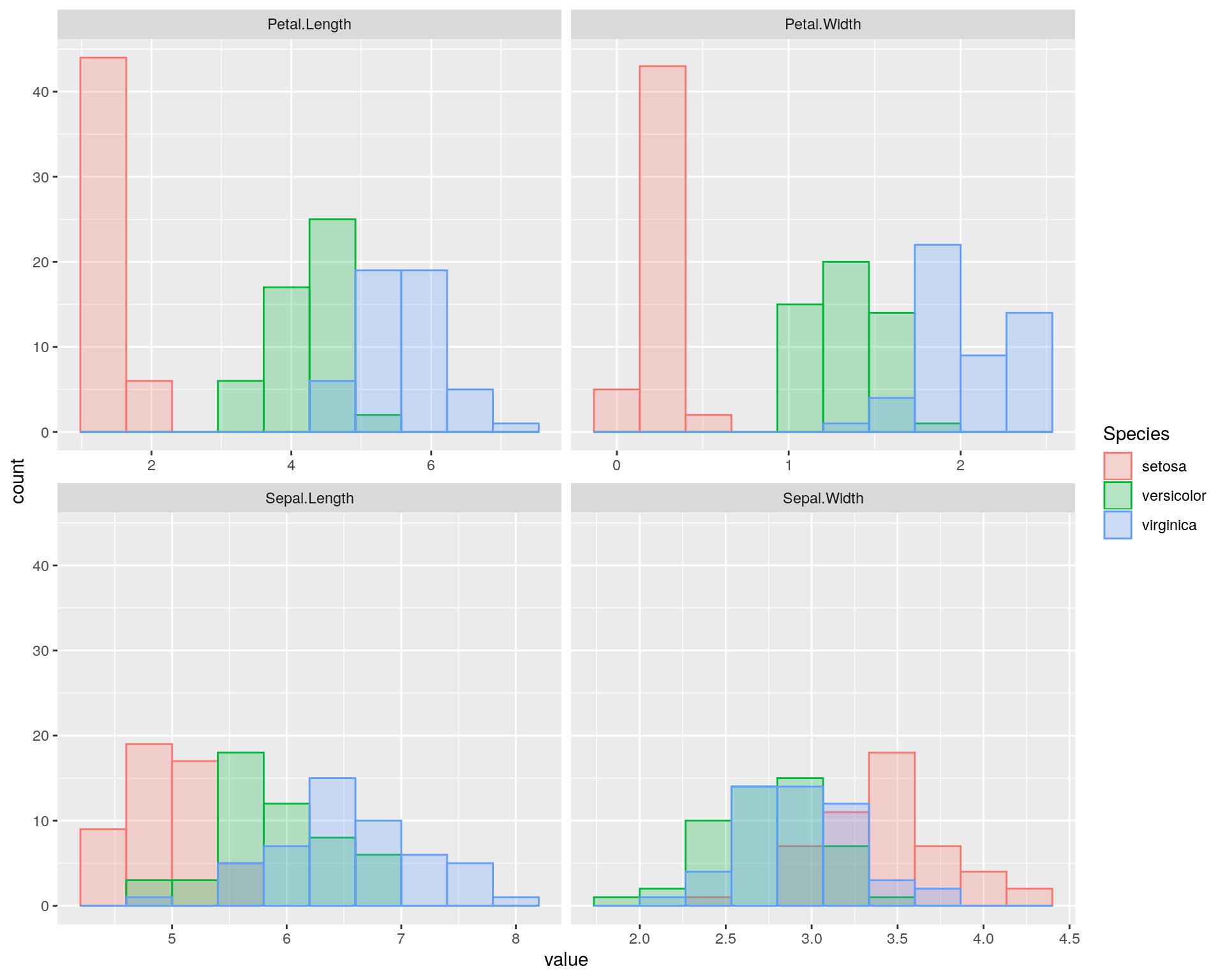
多数の変数を比較する
UC Irvineの機械学習リポジトリにある ウィスコンシン州のがん検診データ には、検査のために採取した細胞の形状に関する数値データが30個、その細胞ががんであるかを判定した結果(因子データ)が1個あります。
wbcd| diagnosis | radius_mean | texture_mean | perimeter_mean | area_mean | smoothness_mean | compactness_mean | concavity_mean | points_mean | symmetry_mean | dimension_mean | radius_se | texture_se | perimeter_se | area_se | smoothness_se | compactness_se | concavity_se | points_se | symmetry_se | dimension_se | radius_worst | texture_worst | perimeter_worst | area_worst | smoothness_worst | compactness_worst | concavity_worst | points_worst | symmetry_worst | dimension_worst |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| B | 12.32 | 12.39 | 78.85 | 464.1 | 0.1 | 0.07 | 0.04 | 0.04 | 0.2 | 0.06 | 0.24 | 0.67 | 1.67 | 17.43 | 0.01 | 0.01 | 0.02 | 0.01 | 0.02 | 0 | 13.5 | 15.64 | 86.97 | 549.1 | 0.14 | 0.13 | 0.12 | 0.09 | 0.28 | 0.07 |
| B | 10.6 | 18.95 | 69.28 | 346.4 | 0.1 | 0.11 | 0.06 | 0.03 | 0.19 | 0.06 | 0.45 | 1.2 | 3.43 | 27.1 | 0.01 | 0.04 | 0.03 | 0.01 | 0.04 | 0 | 11.88 | 22.94 | 78.28 | 424.8 | 0.12 | 0.25 | 0.19 | 0.08 | 0.29 | 0.08 |
| B | 11.04 | 16.83 | 70.92 | 373.2 | 0.11 | 0.08 | 0.03 | 0.02 | 0.17 | 0.06 | 0.2 | 1.39 | 1.34 | 13.54 | 0.01 | 0.01 | 0.01 | 0.01 | 0.02 | 0 | 12.41 | 26.44 | 79.93 | 471.4 | 0.14 | 0.15 | 0.11 | 0.07 | 0.3 | 0.08 |
| NA | … | … | … | … | … | … | … | … | … | … | … | … | … | … | … | … | … | … | … | … | … | … | … | … | … | … | … | … | … | … |
| M | 15.28 | 22.41 | 98.92 | 710.6 | 0.09 | 0.11 | 0.05 | 0.03 | 0.17 | 0.06 | 0.21 | 0.5 | 1.34 | 19.53 | 0 | 0.01 | 0.02 | 0.01 | 0.01 | 0 | 17.8 | 28.03 | 113.8 | 973.1 | 0.13 | 0.33 | 0.36 | 0.12 | 0.32 | 0.1 |
| B | 14.53 | 13.98 | 93.86 | 644.2 | 0.11 | 0.09 | 0.07 | 0.06 | 0.16 | 0.06 | 0.31 | 0.72 | 2.14 | 25.7 | 0.01 | 0.01 | 0.02 | 0.01 | 0.02 | 0 | 15.8 | 16.93 | 103.1 | 749.9 | 0.13 | 0.15 | 0.14 | 0.11 | 0.26 | 0.08 |
| M | 21.37 | 15.1 | 141.3 | 1386 | 0.1 | 0.15 | 0.19 | 0.13 | 0.2 | 0.06 | 0.34 | 1.31 | 2.41 | 39.06 | 0 | 0.03 | 0.03 | 0.01 | 0.02 | 0 | 22.69 | 21.84 | 152.1 | 1535 | 0.12 | 0.28 | 0.4 | 0.2 | 0.27 | 0.09 |
このように多数の変数を持っている場合にはggplot2::facet_wrap関数にncolオプションまたはnrowオプションを指定してグラフが見やすくなるように調整してください。
wbcd %>%
tidyr::gather(key= "vars", value = "value", -diagnosis) %>%
ggplot2::ggplot(ggplot2::aes(x = diagnosis, y = value, color = diagnosis)) +
ggplot2::geom_boxplot() +
ggplot2::facet_wrap(~ vars, scales = "free_y", ncol = 3)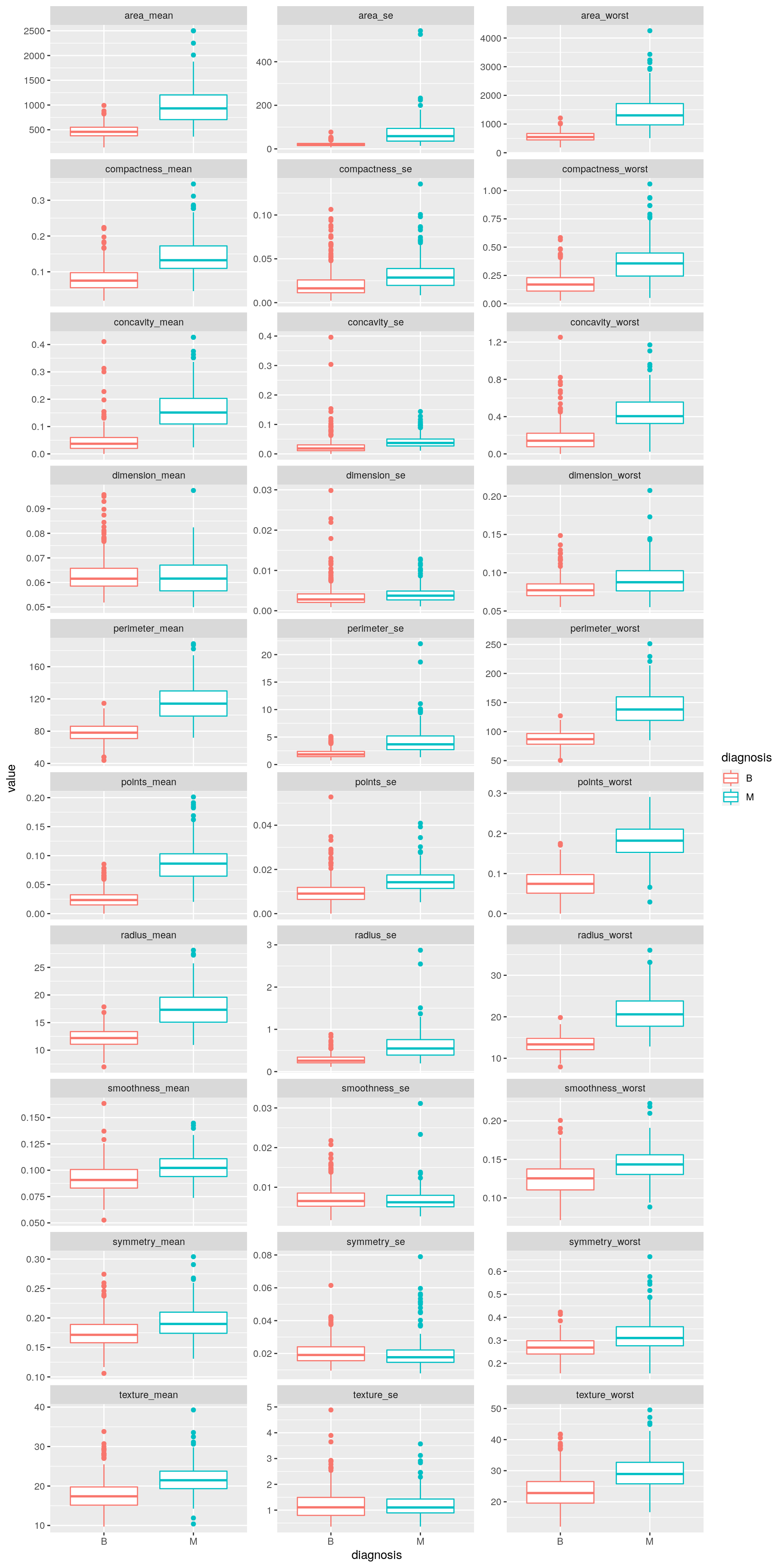
なお、散布図行列を描くことも可能ですが環境によっては数分を要しますのであまり実用的とは言えません。また、利用する関数によってはオーバーフローを起こして描くことができません。
wbcd %>%
GGally::ggpairs(progress = FALSE, ggplot2::aes(colour = diagnosis, alpha = 0.5)) (右クリックメニューで画像だけを表示させれば拡大が可能になります)
(右クリックメニューで画像だけを表示させれば拡大が可能になります)
Enjoy!