Text Update: 11/08, 2018 (JST)
カテゴリカルデータとは質的データや定性的データとも呼ばれ、名義尺度または順序尺度の離散的なデータのことです。データを層別するような場合には大変便利なデータで、 R では因子型変数として扱うことができます。しかし、因子型変数は他の変数とは異なり水準(levels)という属性を持っているために操作が少々厄介な面があります。
Packages and Datasets
本ページではR version 3.4.4 (2018-03-15)の標準パッケージ以外に以下の追加パッケージを用いています。
| Package | Version | Description |
|---|---|---|
| tidyverse | 1.2.1 | Easily Install and Load the ‘Tidyverse’ |
また、本ページでは以下のデータセットを用いています。
| Dataset | Package | Version | Description |
|---|---|---|---|
| iris | datasets | 3.4.4 | Edgar Anderson’s Iris Data |
因子型データ
irisデータセットはSpeciesという因子型変数を持っています。
iris$Species %>% levels()## [1] "setosa" "versicolor" "virginica"
因子型変数は作成時に水準がアルファニューメリック順にソートされますので、グラフ化した場合には下図のようにアルファニューメリック順に表示されます。
iris %>%
ggplot2::ggplot(ggplot2::aes(x = Species, y = Sepal.Width, colour = Species)) +
ggplot2::geom_boxplot()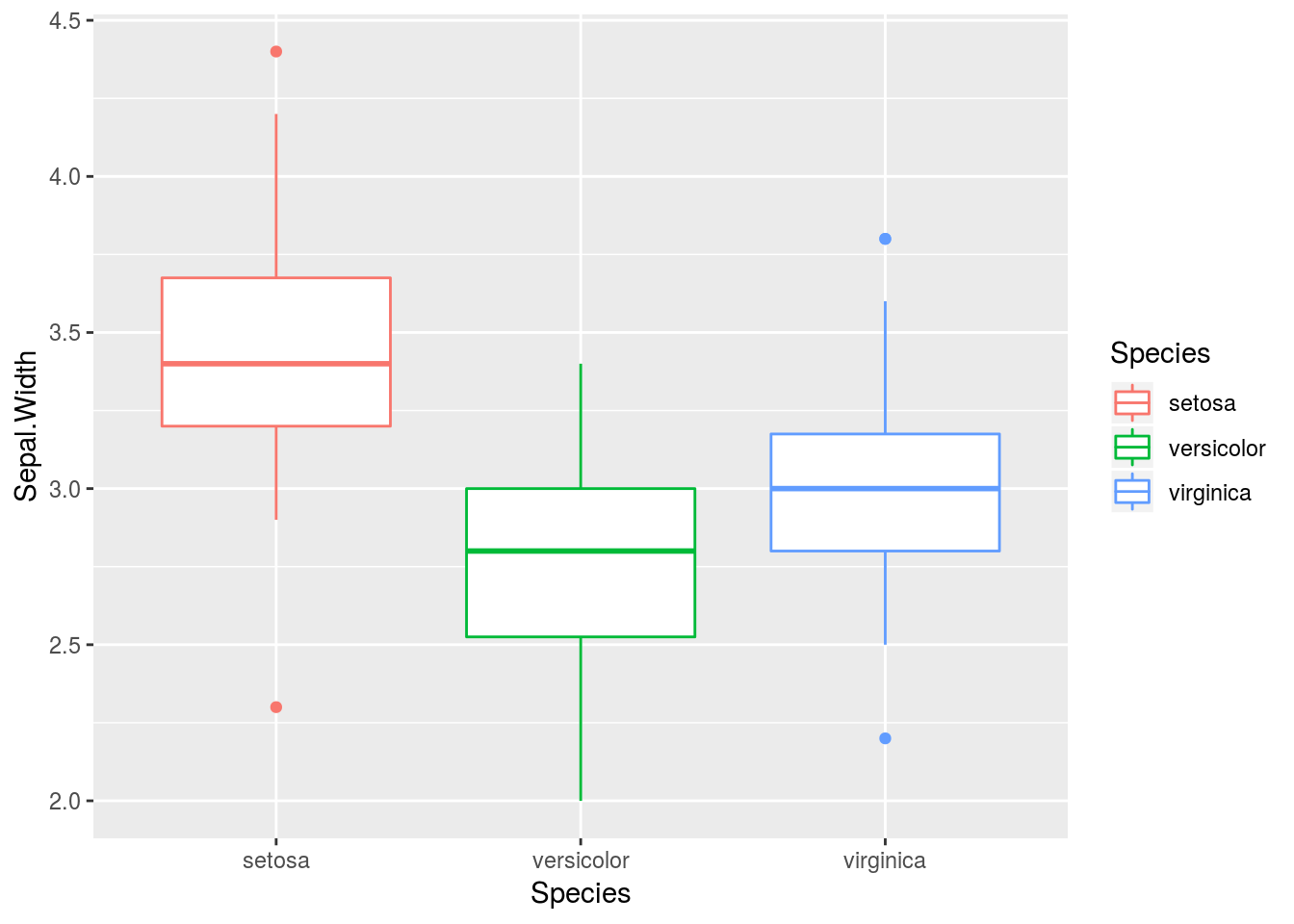
この表示順を変えるには、因子型変数の水準の順番を変更する必要があります。
forcatsパッケージ
因子型変数の水準の順番を変えるような操作を簡単に行えるのがtidyverseファミリーのforcatsパッケージです。tidyverseを読み込むと自動的に読み込まれます。
水準の順番を変える
水準の順番を手動で変えるにはforcats::fct_relevel関数を使います。
iris %>%
dplyr::mutate(Species = forcats::fct_relevel(Species, "versicolor", "virginica")) %>%
ggplot2::ggplot(ggplot2::aes(x = Species, y = Sepal.Width, colour = Species)) +
ggplot2::geom_boxplot()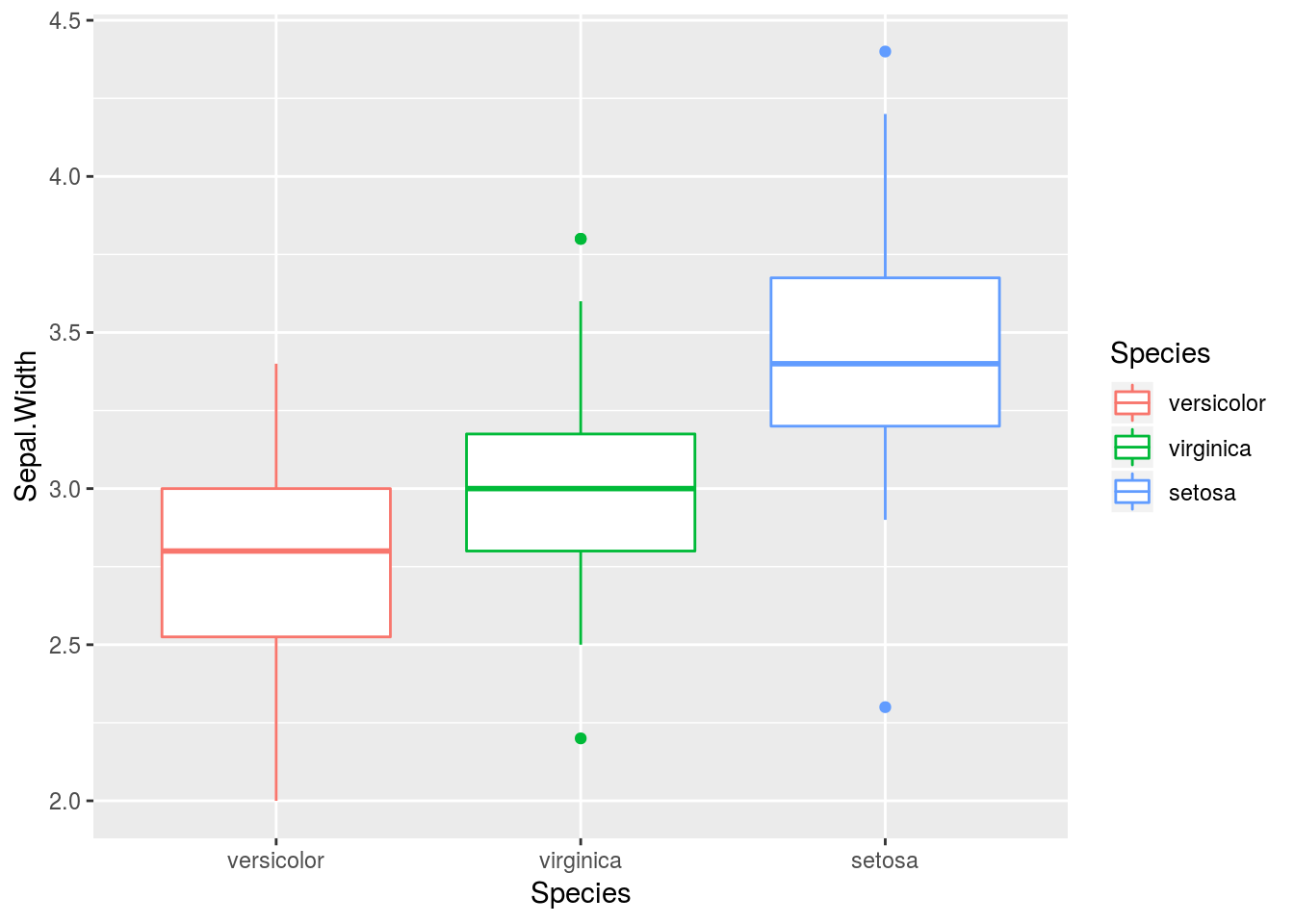
水準の順番を変える
水準の順番を要約統計量を使って並べ変えることも可能です。forcats::fct_reorder関数はデフォルトで指定した変数の中央値で並べ替えます。
iris %>%
dplyr::mutate(Species = forcats::fct_reorder(Species, Sepal.Width)) %>%
ggplot2::ggplot(ggplot2::aes(x = Species, y = Sepal.Width, colour = Species)) +
ggplot2::geom_boxplot()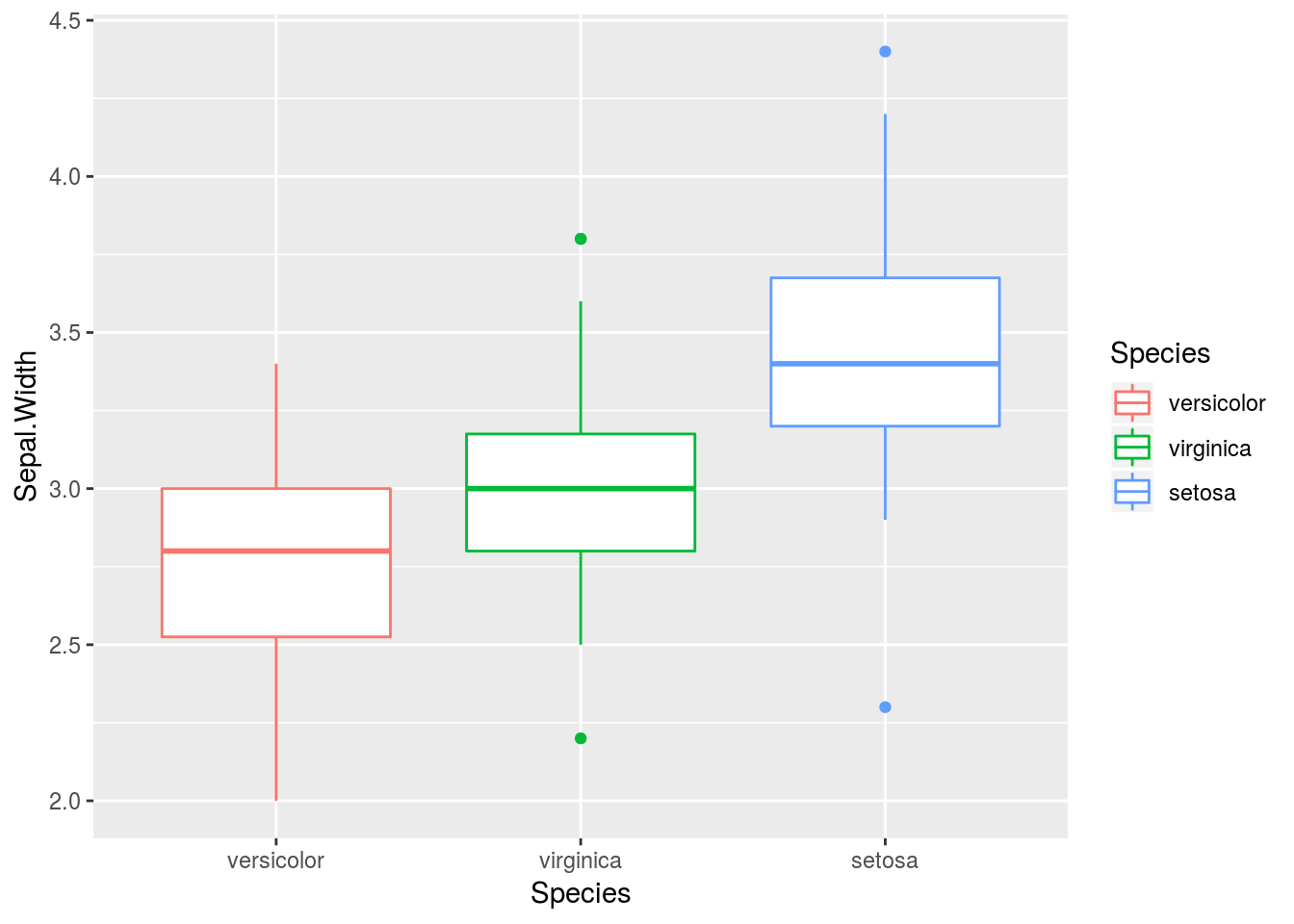
逆順にしたい場合は、forcats::fct_rev関数で並べ直してください。
iris %>%
dplyr::mutate(Species = forcats::fct_reorder(Species, Sepal.Width) %>%
forcats::fct_rev()) %>%
ggplot2::ggplot(ggplot2::aes(x = Species, y = Sepal.Width, colour = Species)) +
ggplot2::geom_boxplot()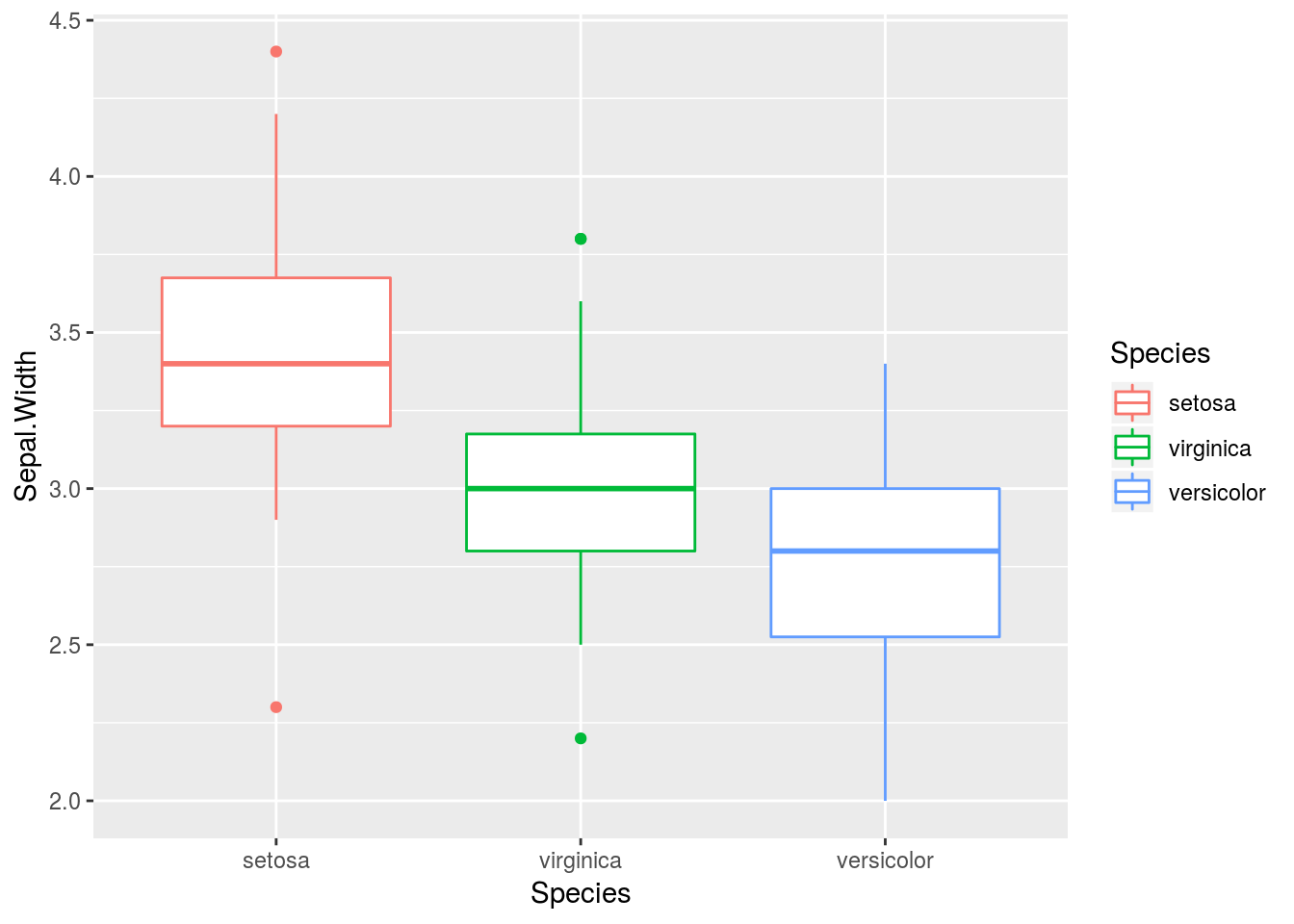
水準を整理する
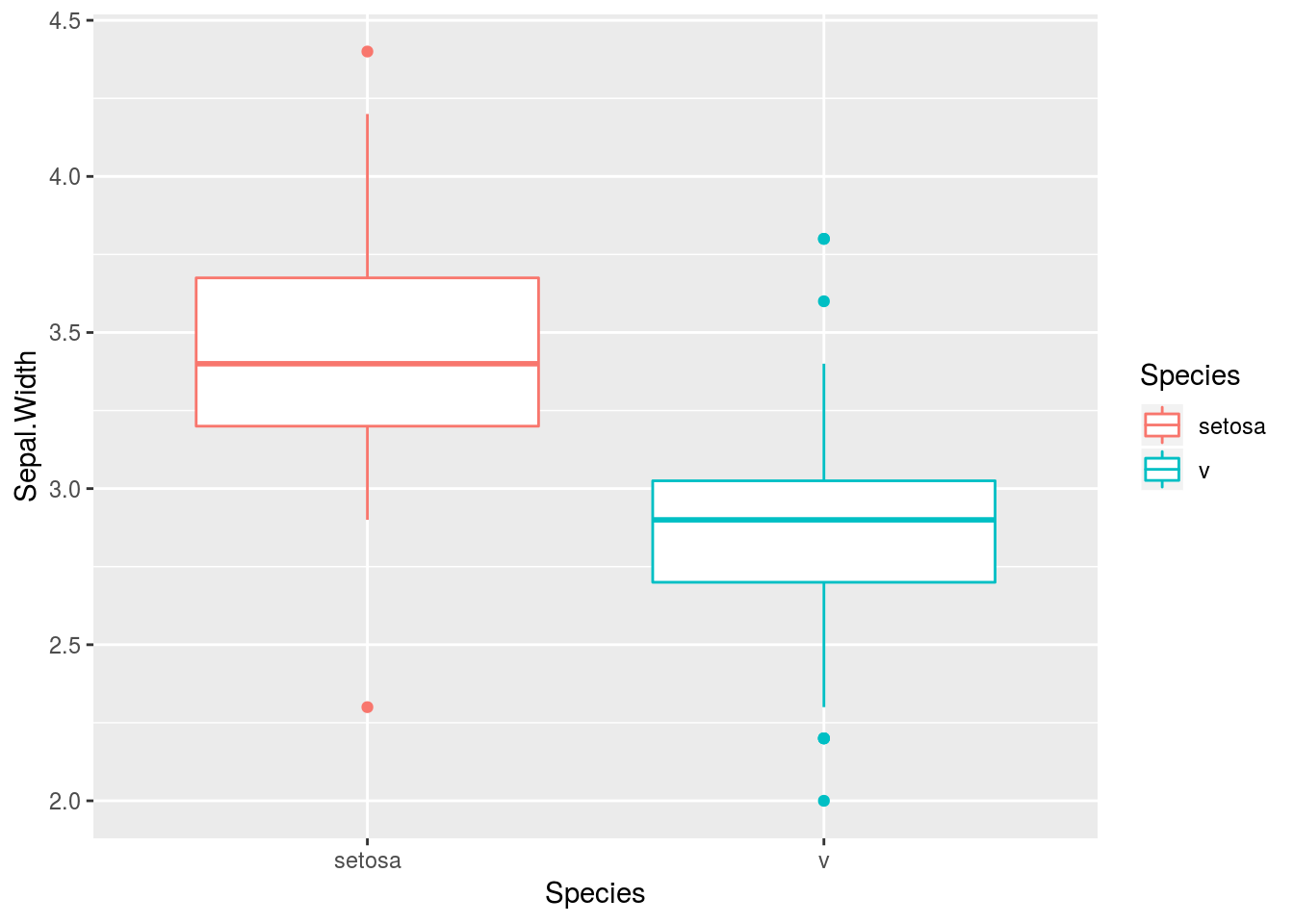
水準をまとめるにはforcats::fct_collapse関数を用います。任意の数だけまとめることが出来ますので、水準数が多すぎる場合などに使ってみてください。
iris %>%
dplyr::mutate(Species = forcats::fct_collapse(Species,
v = c("versicolor", "virginica"))) %>%
ggplot2::ggplot(ggplot2::aes(x = Species, y = Sepal.Width, colour = Species)) +
ggplot2::geom_boxplot()