Text Update: 11/10, 2018 (JST)
データ数が多いグラフやレンジが広いグラフでは値を読み取ることが困難な場合があります。このような場合、インタラクティブに値を表示してくれるグラフを使うと値などが読みやすくなる場合があります。
Packages and Datasets
本ページではR version 3.4.4 (2018-03-15)の標準パッケージ以外に以下の追加パッケージを用いています。
| Package | Version | Description |
|---|---|---|
| plotly | 4.8.0 | Create Interactive Web Graphics via ‘plotly.js’ |
| qqplotr | 0.0.3 | Quantile-Quantile Plot Extensions for ‘ggplot2’ |
| gghighlight | 0.1.0 | Highlight Lines and Points in ‘ggplot2’ |
| tidyverse | 1.2.1 | Easily Install and Load the ‘Tidyverse’ |
また、本ページでは以下のデータセットを用いています。
| Dataset | Package | Version | Description |
|---|---|---|---|
| iris | datasets | 3.4.4 | Edgar Anderson’s Iris Data |
plotly::ggplotly
plotlyパッケージは、パッケージの説明にもあるようにplotly.jsというJavaScriptライブラリによるインタラクティブなグラフを描けるパッケージです。最も簡単な使い方はggplot2オブジェクトをplotly::ggplotly関数に渡すことです。
散布図
ggplot2パッケージで描く散布図は以下のようになります。
iris %>%
ggplot(aes(x = Sepal.Width, y = Sepal.Length, colour = Species)) +
geom_point() +
geom_smooth(method = "lm", se = FALSE)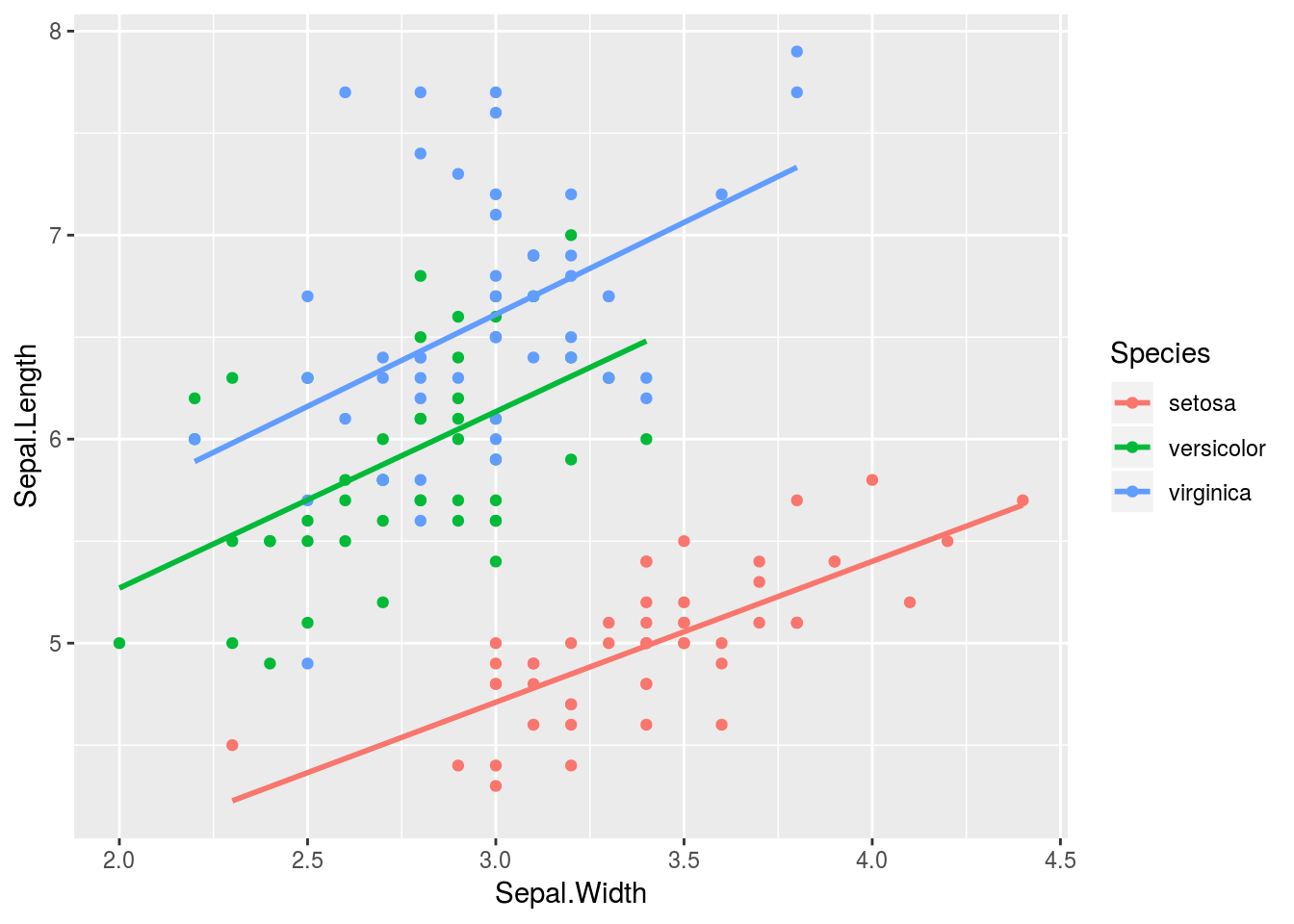
この散布図をそのままplotly::ggplotly関数に渡すとインタラクティブなグラフに変わります。
(iris %>%
ggplot(aes(x = Sepal.Width, y = Sepal.Length, colour = Species)) +
geom_point() +
geom_smooth(method = "lm", se = TRUE)) %>%
plotly::ggplotly()
ヒストグラム
ggplot2パッケージで描くヒストグラムは以下のようになります。
iris %>%
ggplot(aes(x = Petal.Length, fill = Species)) +
geom_histogram(position = "identity", alpha = 0.5)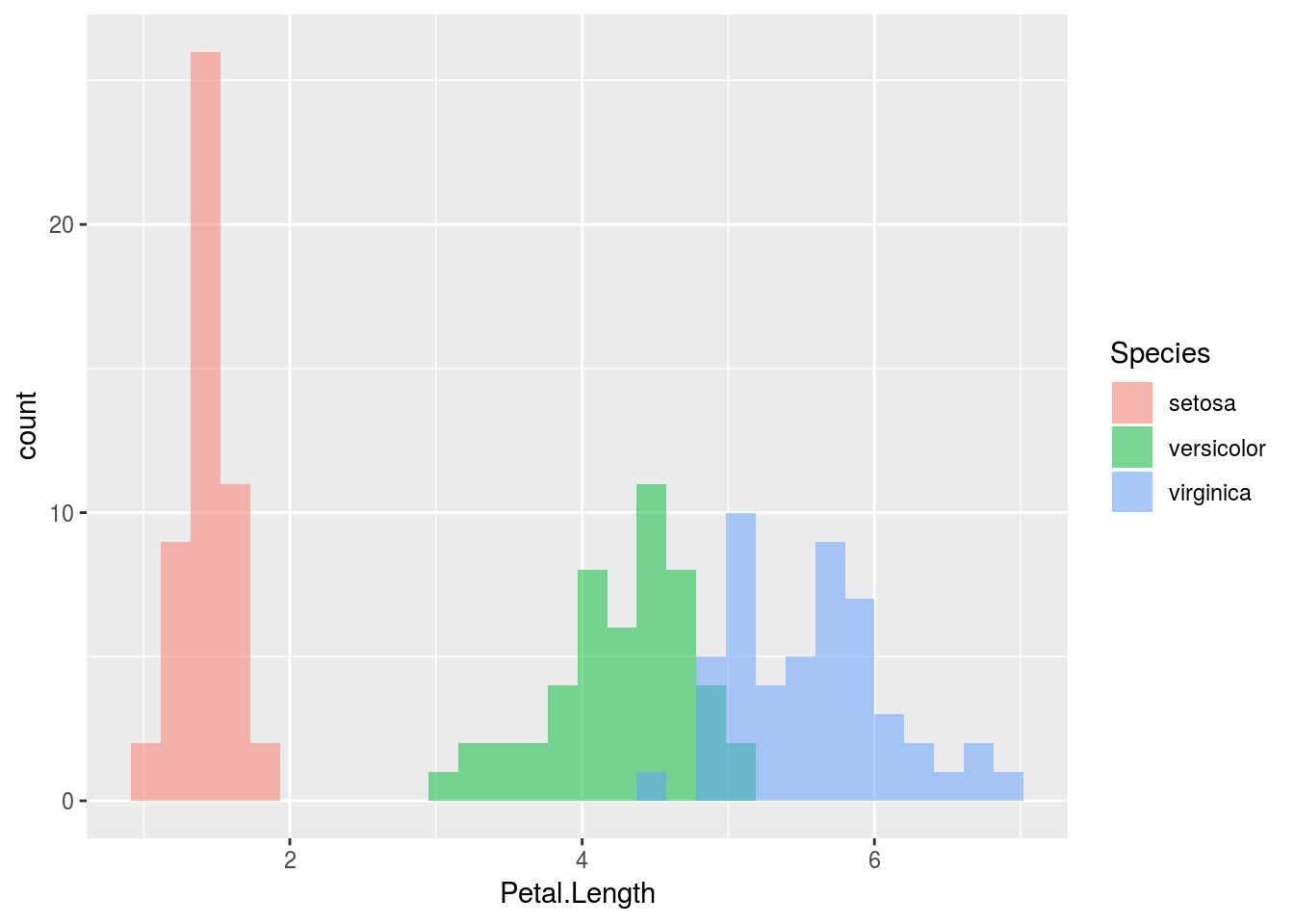
このヒストグラムをそのままplotly::ggplotly関数に渡すとインタラクティブなグラフに変わります。
(iris %>%
ggplot(aes(x = Petal.Length, fill = Species)) +
geom_histogram(position = "identity", alpha = 0.5)) %>%
plotly::ggplotly()箱ひげ図
ggplot2パッケージで描く箱ひげ図は以下のようになります。
iris %>%
ggplot(aes(x = Species, y = Petal.Length)) +
geom_boxplot(aes(colour = Species))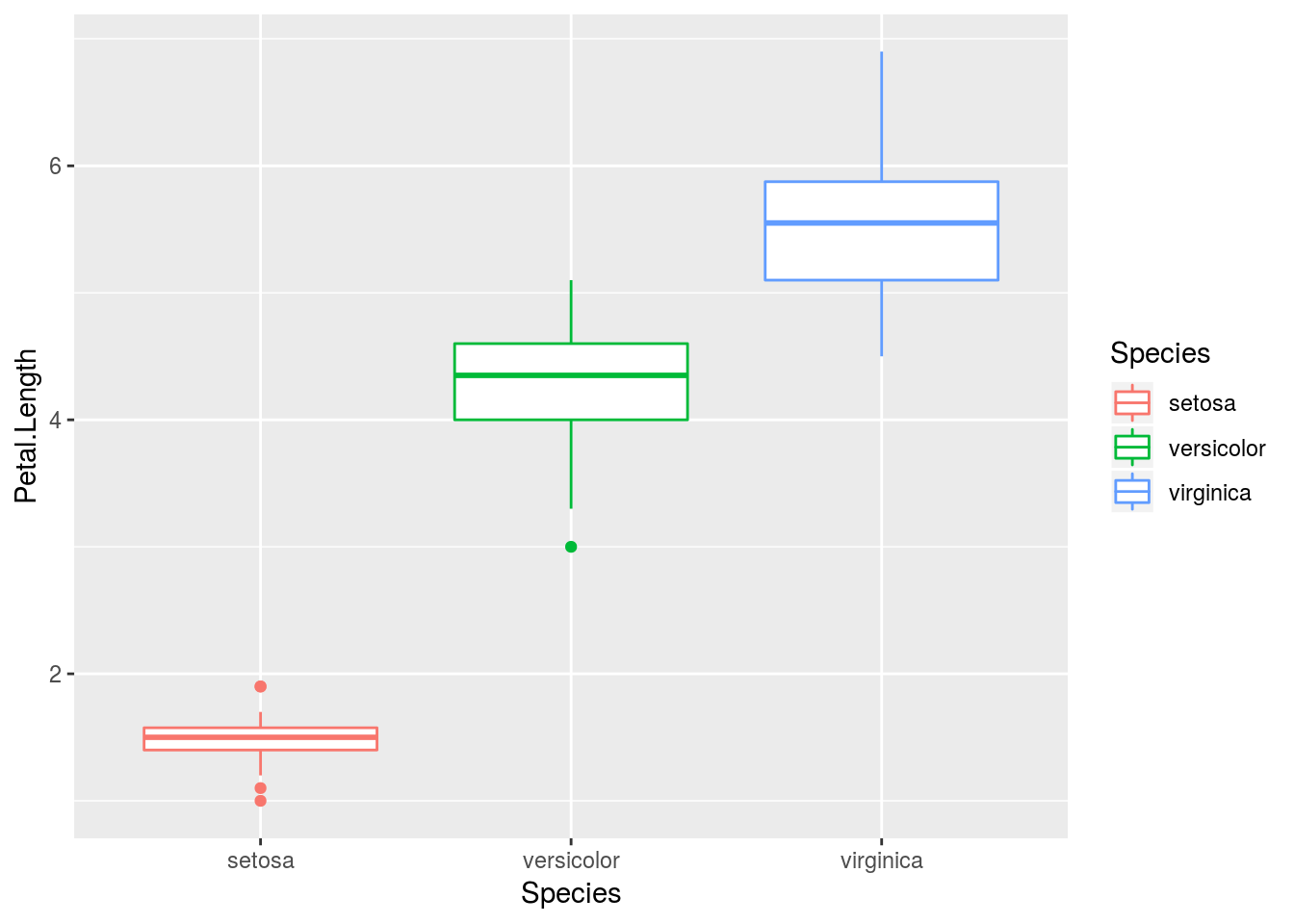
この箱ひげ図をそのままplotly::ggplotly関数に渡すとインタラクティブなグラフに変わります。
(iris %>%
ggplot(aes(x = Species, y = Petal.Length)) +
geom_boxplot(aes(colour = Species))) %>%
plotly::ggplotly()
facet
facet関数群を適用してもインタラクティブなグラフになります。
(iris %>%
ggplot(aes(x = Petal.Width, y = Petal.Length, colour = Species)) +
geom_point() +
geom_smooth(method = "lm", se = FALSE) +
facet_wrap(~ Species)) %>%
plotly::ggplotly()ヘルパーパッケージ
ggplot2のヘルパーパッケージを利用してもパッケージによってはインタラクティブなグラフになります。
(iris %>%
ggplot(aes(Petal.Length, fill = Species)) + facet_wrap(~ Species) +
geom_histogram(position = "identity", alpha = 0.75) +
gghighlight::gghighlight()) %>%
plotly::ggplotly()
このように一部が表示されなくなるケースもあります。
iris %>%
tidyr::drop_na(Sepal.Width) %>%
ggplot2::ggplot(ggplot2::aes(sample = Sepal.Width)) +
qqplotr::stat_qq_band(distribution = "norm",
dparams = list(mean = 0, sd = 1)) +
qqplotr::stat_qq_line(distribution = "norm",
dparams = list(mean = 0, sd = 1)) +
qqplotr::stat_qq_point(distribution = "norm",
dparams = list(mean = 0, sd = 1)) +
ggplot2::theme_minimal() +
ggplot2::labs(x = "Theoretical Quantiles", y = "Sample Quantiles")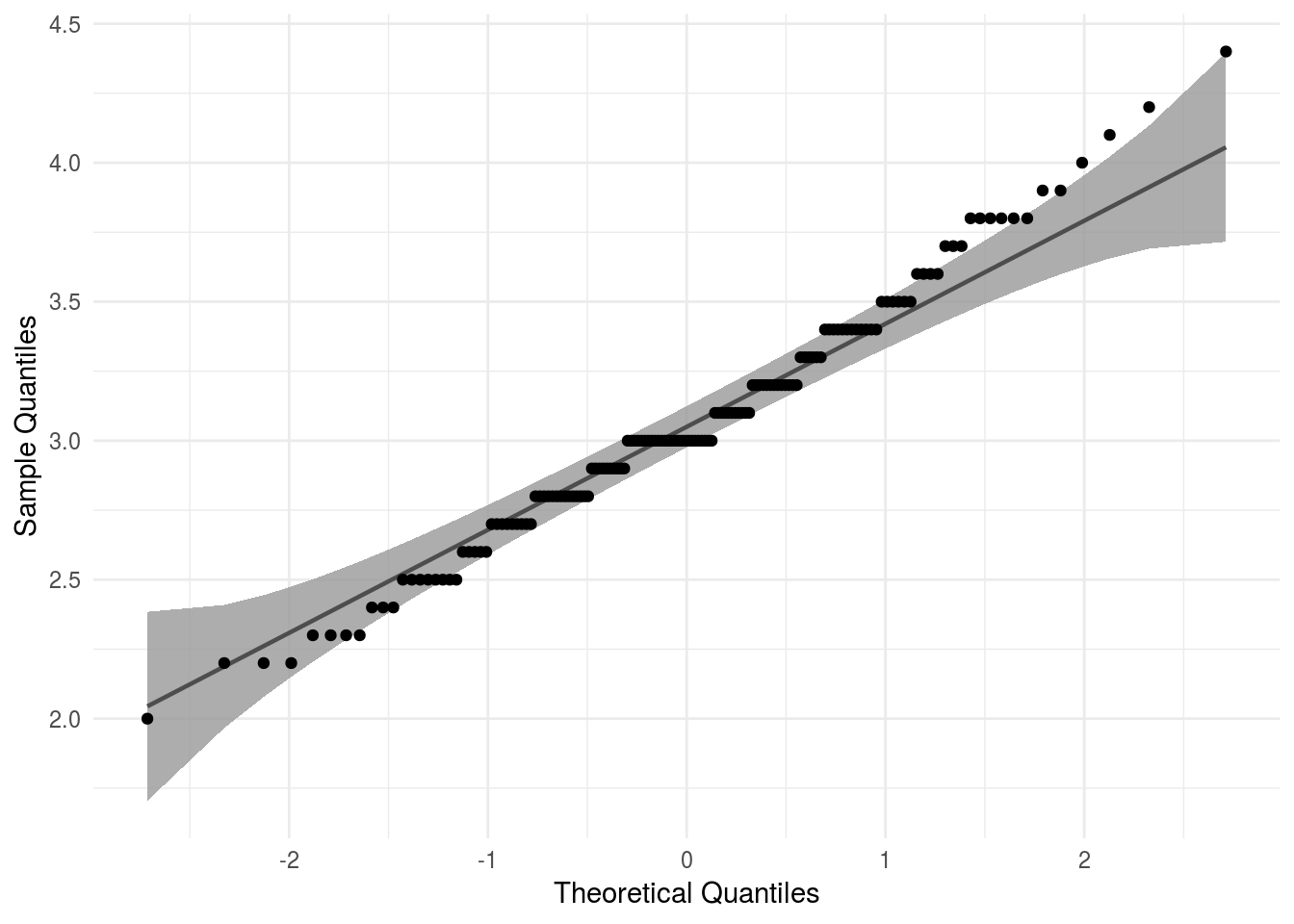
(iris %>%
tidyr::drop_na(Sepal.Width) %>%
ggplot2::ggplot(ggplot2::aes(sample = Sepal.Width)) +
qqplotr::stat_qq_band(distribution = "norm",
dparams = list(mean = 0, sd = 1)) +
qqplotr::stat_qq_line(distribution = "norm",
dparams = list(mean = 0, sd = 1)) +
qqplotr::stat_qq_point(distribution = "norm",
dparams = list(mean = 0, sd = 1)) +
ggplot2::theme_minimal() +
ggplot2::labs(x = "Theoretical Quantiles", y = "Sample Quantiles")) %>%
plotly::ggplotly()
enjoy!